Rスクリプトを使って、フォルダ内のcsvやエクセルを
data.tableパッケージののfreadと、readxlパッケージのread_excelを使って
freadの場合は
lf <- list.files(path = “フォルダのパス”, full.names = T,pattern ="*.csv")
data <- lapply(lf,fread,sep=’,’, colClasses=“character”)
data_bind <- do.call(rbind, data)
read_excelの場合は
lf <- dir(path = “フォルダのパス”,full.names = T,pattern=".xlsx")
data <- do.call(rbind, lapply(lf,read_excel,col_types=“text”))
というようなかたちで一気に取り込んでいますが、
このときに取り込み元のファイル名を列項目として入れておきたいのですが、
なにかよい方法はないでしょうか。
【追加】
すいません。文意を取り違えていました。
別々の拡張子のデータを一度に同じデータとして取り込むわけではないようですね。
それであれば下記だけで問題ないと思います。
setwd()でフォルダまでのパスを設定しておけば、
不要なパスを取り除く処理も必要なくなりますし。
Excelの場合も同様です。
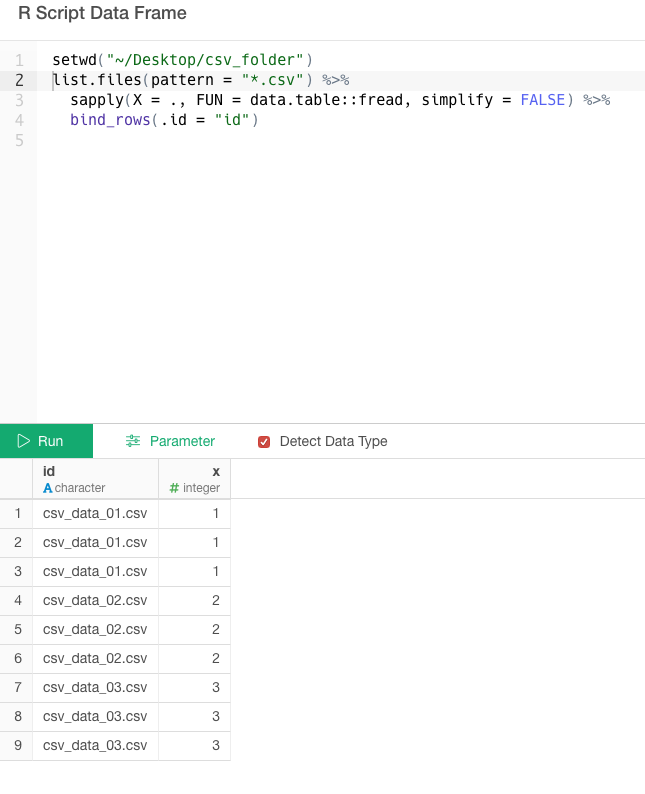
setwd("~/Desktop/csv_folder")
list.files(pattern = "*.csv") %>%
sapply(X = ., FUN = data.table::fread, simplify = FALSE) %>%
bind_rows(.id = "id")
Yasuhiro_Yatsuu さま
はじめまして。
下記のように読み込むのはいかがでしょうか。
folder_csv、folder_xlsxには3つづデータが入っている想定です。
{data.table}を使われているということはそれなりにサイズの大きいCSVだと思いますが、
ここではread_excel()が返す型をあわせるためにread_csv()を使用してます。
fread()だとdata.tableかdata.frameしか返せないのでこうしました。
また、ちょっといいやり方が思いつかず、データ名をとりたいがために、
無理やりこのようにしてとっていますが、unest()が読み込むデータサイズによっては、
もしかすると遅くなる要因となるかも。
library(fs)
library(readr)
library(purrr)
library(stringr)
library(tidyr)
library(dplyr)
library(readxl)
dir_csv <- list.files(path = "C:/Users/<user.name>/Desktop/folder_csv", full.names = TRUE, pattern = "*.csv")
dir_xlsx <- list.files(path = "C:/Users/<user.name>/Desktop/folder_xlsx", full.names = TRUE, pattern = "*.xlsx")
dir <- c(dir_csv, dir_xlsx)
tibble(dir) %>%
mutate(data = map(.x = dir,
.f = function(x){
if(path_ext(x) == "csv"){
read_csv(x)
} else {
read_excel(x)
}
}),
dir = str_extract(string = dir, pattern = "(?<=/)(?!.*/).+")) %>%
unnest(data)
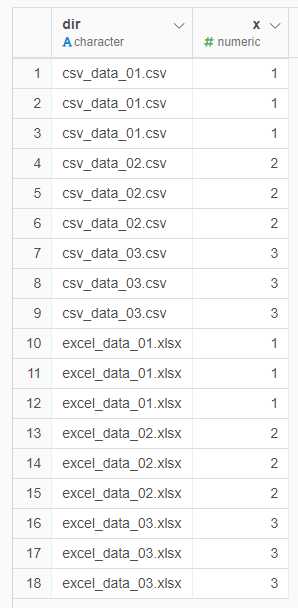
下記のような方法でも同様のことはできるかと思います。
library(stringr)
library(dplyr)
library(readxl)
library(data.table)
dir_csv <- list.files(path = "C:/Users/<user.name>/Desktop/folder_csv",
full.names = TRUE,
pattern = "*.csv")
df_csv <- sapply(X = dir_csv,
FUN = data.table::fread,
simplify = FALSE) %>%
bind_rows(.id = "id")
dir_xlsx <-list.files(path = "C:/Users/<user.name>/Desktop/folder_xlsx",
full.names = TRUE,
pattern = "*.xlsx")
df_xlsx <- sapply(X = dir_xlsx,
FUN = readxl::read_excel,
simplify = FALSE) %>%
bind_rows(.id = "id")
df_xlsx %>%
bind_rows(., df_csv) %>%
mutate(dir = str_extract(string = id, pattern = "(?<=/)(?!.*/).+"))

よろしくお願いいたします!
「いいね!」 3
ありがとうございます。
うっとりするぐらい素晴らしいです。
たいへん勉強になります。
パイプ演算子を使っているのは、
bind_rowsの.idでlist.filesの名前を取ってくるのを
lapplyなりsapply(の中でやるのはちょっと辛そう)の外側でやるため、
と理解しましたが認識相違ないでしょうか。
Yasuhiro_Yatsuuさま
パイプ演算子を使っているのは、
私のコーディングスタイルによるものと、
Exploratory自体がTidyverseの世界観を踏まえている(個人的な考え)ためで、
特に意味はありません・・・
なので、下記のようにパイプを使わない方法でも実現できます。
setwd("C:/Users/<username>/Desktop/csv_folder")
target_csv <- list.files(pattern = "*.csv")
target_csv_read <- sapply(X = target_csv, FUN = read_csv, simplify = FALSE)
bind_rows(target_csv_read, .id = "id")
# A tibble: 6 x 2
id x
<chr> <dbl>
1 csv_data_01.csv 1
2 csv_data_01.csv 1
3 csv_data_01.csv 1
4 csv_data_02.csv 2
5 csv_data_02.csv 2
6 csv_data_02.csv 2
lapply()の代わりにsapply()を使っている点の方が理由がありまして、
sapply()だと受け取ったデータの値をリストの名前として付与できますが、
lapply()だと連番になってしまいます。
データの識別IDのようなものを付与できれば、それでも問題ということであればよいのですが、
今回はデータ名が欲しいという内容でしたので、sapply()を使っています。
また、もちろん、やろうと思えば、lapply()なりsapply()‘の中で何とかすることは可能かと思いますが、 bind_rows() の .id 引数を使えば、sapply()からの流れで、不要な処理やコードの可読性をさげることなく、
やりたいことが実現できるため、このような感じになっています~。
「いいね!」 1
lapply()の代わりにsapply()を使っている点の方が理由がありまして、
sapply()だと受け取ったデータの値をリストの名前として付与できますが、
lapply()だと連番になってしまいます。
結果が「リスト」か「ベクトル」か、ぐらいの知識しかなかったので、
これは大変に勉強になりました!
ありがとうございます!
「いいね!」 1
少しばかりですが、お力添えできたようで、よかったです!
私もまだまだ知らないことが多いですが、一緒に頑張りましょう!
引き続きよろしくお願いいたします~!


